- 세포 염색 이미지와 단백질 서열 결합⋯AI로 단일 세포 수준 정밀 예측
- 질병 진단·신약 개발 가속 기대⋯"실험 없이 가상 예측 가능"
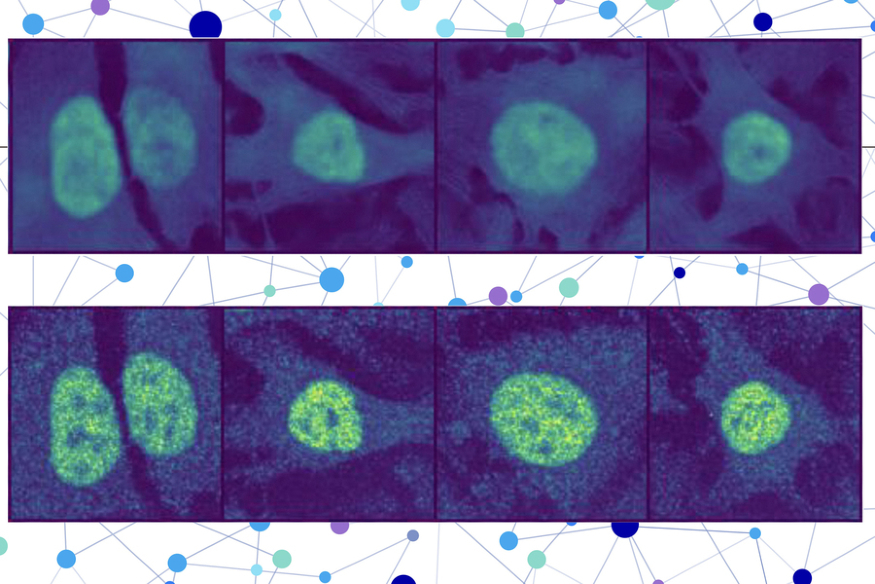
미국 MIT, 하버드대, 브로드연구소 공동 연구팀이 인공지능(AI)을 활용해 인간 세포 내 거의 모든 단백질의 위치를 예측할 수 있는 신기술을 개발했다. 이 기술은 단백질 이상 분포가 원인이 되는 알츠하이머병, 낭포성 섬유증, 암 등 다양한 질환 진단과 신약 개발에 폭넓게 활용될 것으로 기대된다.
15일(현지시간) MIT뉴스에 따르면 이번 연구는 단일 세포 수준에서 단백질 위치를 예측할 수 있는 AI 기반 모델 'PUPS(Protein localization Using Protein and cell representationS)'를 중심으로 소개됐다.
해당 연구는 15일 국제 학술지 네이처 메서즈(Nature Methods)'에 게재됐다.
단일 세포 내 단백질 분포 예측⋯기존 한계 돌파
단백질은 세포 내 위치에 따라 기능이 달라지며, 잘못된 위치에 존재할 경우 각종 질병을 유발할 수 있다. 하지만 인간 세포 하나에 존재하는 단백질과 그 변이 수는 7만여 개에 달하며, 실험실에서 이들을 일일이 검출하는 데는 시간과 비용이 과도하게 소요된다.
기존의 AI 기반 예측 모델들은 훈련 데이터로 활용된 단백질 및 세포에 국한된 분석만 가능하거나, 세포 단위 평균치만을 예측하는 데 그쳤다. 이에 반해 PUPS는 개별 단백질 서열 정보와 세포 염색 이미지를 결합해, 실험에 사용된 적이 없는 단백질과 세포에서도 단일 세포 수준의 위치 예측이 가능하다는 점에서 큰 진전을 이뤘다.
단백질 언어모델+영상 AI 결합⋯미지 영역 탐색
PUPS는 두 개의 AI 모델을 결합한 형태로 구성됐다. 첫 번째는 단백질 서열 기반 언어모델로, 아미노산 서열을 통해 3차원 구조와 세포 내 위치 결정에 영향을 미치는 특성을 분석한다. 두 번째는 '이미지 인페인팅(image inpainting)' 기법을 적용한 컴퓨터 비전 모델로, 세포의 핵, 미세소관, 소포체 등 세 가지 염색 이미지를 바탕으로 세포의 형태, 스트레스 상태 등 전반적 특성을 파악한다.
연구진은 이 두 모델이 생성한 정보 표현을 통합해 특정 단백질이 어느 세포 소기관에 위치할지를 시각적으로 예측하는 시스템을 구현했다. 사용자 입장에서는 아미노산 서열과 세포 염색 이미지를 입력하면, 단백질이 존재할 것으로 예상되는 세포 부위를 이미지로 출력받을 수 있다.
단백질-세포 간 조합 없이도 예측 가능⋯신규 치료 타깃 발굴 기대
PUPS의 강점은 기존 데이터에 포함되지 않은 단백질과 세포 간 조합에도 적용할 수 있다는 점이다. 이를 위해 연구진은 모델 학습 시 단백질의 위치를 예측하는 주된 작업 외에, 단백질이 속한 소기관의 이름을 직접 추론하도록 하는 ‘부가 학습 과제’를 부여해 정확도를 높였다.
공동 제1저자인 MIT 시스템생물학 대학원생 Yitong Tseo는 "이전에는 단백질에 직접 염색 시약을 붙이고 실험을 해야만 위치를 알 수 있었지만, 이제는 연구실 벤치에 손대지 않고도 그 과정을 컴퓨터에서 선별적으로 예측할 수 있다"며, 실험 설계의 효율성을 크게 높일 수 있다고 밝혔다.
실제 세포 실험으로 검증…"기존 AI보다 오차 작아"
연구팀은 PUPS의 예측 결과가 실제 단백질 위치와 얼마나 일치하는지를 검증하기 위해 실험실 실험을 병행했다. 비교 대상이 된 기존 AI 모델보다 평균적으로 오차가 적은 것으로 나타났다.
향후 연구진은 PUPS가 단일 단백질이 아닌 다중 단백질 상호작용을 반영할 수 있도록 모델을 확장할 계획이며, 궁극적으로는 배양 세포를 넘어 실제 조직 단위의 예측으로 발전시킬 수 있도록 연구를 지속할 방침이다.
이번 연구는 브로드연구소 산하 에릭 앤 웬디 슈미트 센터와 미국 국립보건원(NIH), 국립과학재단(NSF), 해군연구청(ONR), 에너지부(DOE) 등 주요 기관의 지원을 받아 진행됐다.






